Table of Contents
Commit 2017-03-13
Modèles de profilage
Contexte
Ce commit marque essentiellement l’aboutissement des nouveaux développements que j’ai apportés à nos modèles numériques de profilage dans le cadre du papier IJSS. L’objectif premier poursuivi dans ce papier est la modélisation de l’enchaînement de procédés utilisés en aval du procédé de profilage, à savoir la soudure en ligne qui ferme le produit mis en forme et ensuite la découpe en sortie machine afin de libérer le profilé des outillages et ainsi obtenir sa géométrie finale. La première étape est la prise en compte de la soudure en ligne au travers des sticking elements spécifiquement implémentés (voir mon précédent commit) dans le calcul ALE du profilage. La seconde partie consiste en une désactivation des éléments finis de la bande à l’intérieur de la machine (voir ce commit).
Cas-tests de profilage
Les trois cas-tests de profilage mis au point pour le papier IJSS sont ajoutés dans la batterie de tests. Ceux-ci sont chargés sans réaliser l’intégration temporelle. De plus, des cas-tests extrêmement réduits (limités à une seule station de mise à forme) sur base de ces précédents gros cas-tests sont inclus dans la batterie. Pour ceux-ci, l’enchaînement du profilage avec collage des rives et découpe est simulé.
Répertoire rollForming/copraCpeCre/
- Modifications de la ligne de profilage d’un tube cylindrique (Tube.CPE/.CRE) : ajout d’un premier profil parfaitement plat et d’une première station sans outillage.
- Ajout de la version originelle de la ligne de profilage du tube (Tube.orig.CPE/.CRE).
CopraImportUtilities.py
- L’utilisateur peut définir des conditions aux limites en un nœud de son choix des sections droites amont et aval (utilisé dans le cas de la post-découpe Lagrangienne d’un profil fermé).
- Une liste de stations peut désormais être supprimée.
- Possibilité d’élargissement du contour d’un outil (copraRoll) si l’entité à l’extrémité du contour est un Arc (utilisé pour le cas-test du profilage du tube).
- Possibilité d’écrire un fichier Matlab décrivant la connectivité du maillage.
- La définition de la fonction
printParameters2Matlab(param)est supprimée puisque celle-ci est à présent définie danstoolbox/matlab.py.
copraRollForming.py
- Ajout d’une option pour le calcul parallèle de la détection du contact.
- Possibilité d’utiliser le schéma d’intégration alpha-généralisé amorti pour le calcul de la phase finale du retour élastique.
- Le fichier Matlab des paramètres du cas-test est désormais écrit en toute fin du calcul avec une
ObjectiveFunction.
copraRollFormingALE.py
- Ajout d’une option pour le calcul parallèle de la détection du contact.
- Le fichier Matlab des paramètres du cas-test est désormais écrit en toute fin du calcul avec une
ObjectiveFunction. - Possibilité de modéliser un collage en ligne pour les sections fermées. Collage par
sticking elementou par conditions aux limites de symétrie. - Possibilité d’un nombre d’éléments finis différent pour chacun des plis.
- Possibilité d’élargir les zones de raffinement du maillage par un facteur différent pour chacun des plis.
- Possibilité de contrôler le raffinement du maillage en rives.
- Possibilité d’utiliser le schéma d’intégration alpha-généralisé amorti pour le calcul de la phase Lagrangienne finale du retour élastique après découpe.
- Ajout de paramètres de contrôle de la taille du pas de temps.
- Ajout d’un paramètre de contrôle du nombre de mailles fixées dans le groupe en entrée de ligne.
- Sélection des nœuds milieux des profils amont et aval plus robuste.
- Possibilité d’ajouter des interactions de contact spécifiques (exemple du profilage du tube symétrique : contact entre le plan de symétrie et la rive).
- La convection de
IF_RUPT_CRITest ignorée. - Ajout d’options pour contrôler les méthodes de flux.
- Possibilité de remailler les rives à coordonnées curvilignes constantes ou bien à coordonnées cartésiennes constantes.
importCopra.py
La fonction importCopraFiles(testName) renvoie le nom du fichier et non plus le nom du module.
materialDataBase.py
Ajout d’un modèle de matériau dont les paramètres sont définis ici.
utilitiesCopra.py
Correction de petits bugs dans les outils de preprocessing :
- La classe
RollFormingLinen’a plus de méthodegetParameters()depuis la révision 2570. - Prise en compte dans le dictionnaire de paramètres de l’épaisseur de bande définie dans le fichier .CPE si l’utilisateur ne l’a pas définie.
marc2py.py
Depuis que les éléments 0D sont générés au travers de Mechanism0DInteraction, il n’était plus possible de lancer l’intégration temporelle d’un cas-test Lagrangien de profilage industriel dans lequel le maillage est importé à partir du code Marc FEA. Pour ce cas-test, seul le chargement du modèle est testé dans la batterie, sans aucune intégration temporelle, le problème ne pouvait être mis en évidence par la batterie. L’interaction problématique repose sur un groupe défini comme ce qui suit :
volume = groupset.add(Group(idx+100)) mesh.define(idx+1,CELL_HEXA,volume,[tp(idx+1),tp(idx+117),tp(idx+119),tp(idx+3),tp(idx+2),tp(idx+118),tp(idx+120),tp(idx+4)]) mesh.define(idx+2,CELL_HEXA,volume,[tp(idx+3),tp(idx+119),tp(idx+121),tp(idx+5),tp(idx+4),tp(idx+120),tp(idx+122),tp(idx+6)]) mesh.define(idx+3,CELL_HEXA,volume,[tp(idx+5),tp(idx+121),tp(idx+123),tp(idx+7),tp(idx+6),tp(idx+122),tp(idx+124),tp(idx+8)]) mesh.define(idx+4,CELL_HEXA,volume,[tp(idx+7),tp(idx+123),tp(idx+125),tp(idx+9),tp(idx+8),tp(idx+124),tp(idx+126),tp(idx+10)]) ...
Lors de la construction de l’interaction, le groupe ne comportait aucun meshPoint, ce qui générait une erreur fatale. Finalement, pour résoudre ce problème, j’ai ajouté dans le groupe en question les meshPoints du maillage importé.
Imports relatifs
Dans rollForming/copraTools, tous les imports absolus sont remplacés par des imports relatifs, à l’exception de l’import dans le fichier Python décrivant la ligne de profilage (traduit à partir des fichiers .CPE/.CRE) qui reste un import absolu.
workspace des cas-tests par enchaînement
Pour tous les cas-tests de profilage par enchaînement (une phase de profilage suivie d’une phase de calcul du retour élastique en dehors de la machine), la seconde phase du calcul est dès à présent réalisée dans un sous-répertoire de travail différent de celui de la première pour autant que cette première phase ne se soit pas achevée sur une configuration non-équilibrée (détection du nom de fichier step_neq.bfac.gz). Si la première phase du calcul s’achève sur une configuration non-équilibrée, la seconde phase du calcul n’est désormais plus démarrée automatiquement.
Réorganisation des cas-tests de profilage
- Réorganisation des répertoires
oo_nda/rollForming/etoo_nda/montataire/comme déjà réalisée dansoo_nda/crm/(voir ce commit), c'est-à-dire que tous les cas-tests spécifiques au système de batterie sont dès à présent regroupés dans un seul répertoire dédié. Les différents choix (meta(),prepro(), import du jeu de données) dans la fonctionmain()des gros cas-tests sont ainsi supprimés. - Ajout du répertoire
batterydansoo_nda/montataire. - Ajout du répertoire
copraBatterydansoo_nda/rollForming.
⇒ Modification des CMakeLists.txt respectifs.
- Move de différents cas-tests.
- Ajout de $Id$ dans l'en-tête des fichiers.
toolbox/abaqus.py
Petite correction sur la position courante des nœuds.
toolbox/matlab.py
Ajout d’un export de la connectivité du maillage.
toolbox/utilities.py
Depuis la révision 2096, la base du répertoire de travail wDirRoot avait été ajoutée dans le path pour permettre de charger le fichier Python décrivant les outillages et les profils de la ligne de profilage sur les disques locaux de Fabulous. Désormais, cet ajout est commentarisé. Le fichier Python est importé à partir de son nom avec les méthodes imp.find_module(impMod, [buildWDir]) et imp.load_module(impMod, file, pathName, description) et non plus avec la méthode import(moduleName, globals(), locals(),[moduleName] ) qui nécessite le nom du module comportant son chemin.
Renaming dans mtDrawables
Les classes SFScalars et SFVectors nécessaires à la visualisation des champs scalaire et vectorielle des interactions de collage sont renommés StickFScalars et StickFVectors respectivement pour une plus grande clarté.
mtALE/SplineCartCurveReZoner
Avec le reZoner à coordonnées cartésiennes constantes de mon précédent commit, des problèmes de convergence dans le repositionnement des nœuds sont survenus dans les simulations du profilage, avec pour conséquence des divisions du pas de temps. Ces problèmes de convergence n’avaient pas été rencontrés dans les petits cas-tests de collage de mon précédent commit, ni dans mes simulations de profilage pour le papier IJSS. Entretemps, j’avais réécrit l'algorithme du calcul de l’intersection entre une spline et un plan à coordonnées cartésiennes constantes avec un Newton-Raphson en lieu et place d’une méthode de la sécante. Pour résoudre ces problèmes de convergence, j’ai modifié le premier guess dans le Newton-Raphson.
mtGeo/ProfileBuilder
Après avoir réorganisé le module des modèles de profilage, j’ai lancé la batterie de tests pour ce module en particulier (-run rollForming), ce qui a entraîné, outre le lancement des simulations du répertoire rollForming/copraBattery/, le lancement inattendu des simulations présentes dans le répertoire rollForming/copraTests/ (gros cas-tests). Pis, parmi ces dernières, j’ai pu observer que l’une s’est achevée d’emblée sur une erreur fatale en raison d’un jacobien négatif, avant même de calculer le premier pas de temps. Dans la procédure classique de la batterie, ce cas-test est chargé mais sans aucune intégration temporelle. Le système de batterie ne pouvait dès lors faire apparaître ce cas-test comme failed.
Tout d’abord, observer le problème dans le maillage ALE ne fut pas évident vu la taille considérable du modèle. J’ai commencé par déterminer la demi-partie du profil concernée par le problème. Ensuite, j’ai identifié l’inter-tête du maillage en réduisant le nombre de stations importées dans le modèle. De cette manière, j’ai abouti au modèle réduit illustré ci-après. On y observe un défaut dans la géométrie des coins de la rive. La construction des deux polyCurves frontières à partir de la polyCurve à mi-épaisseur paraît être en cause.
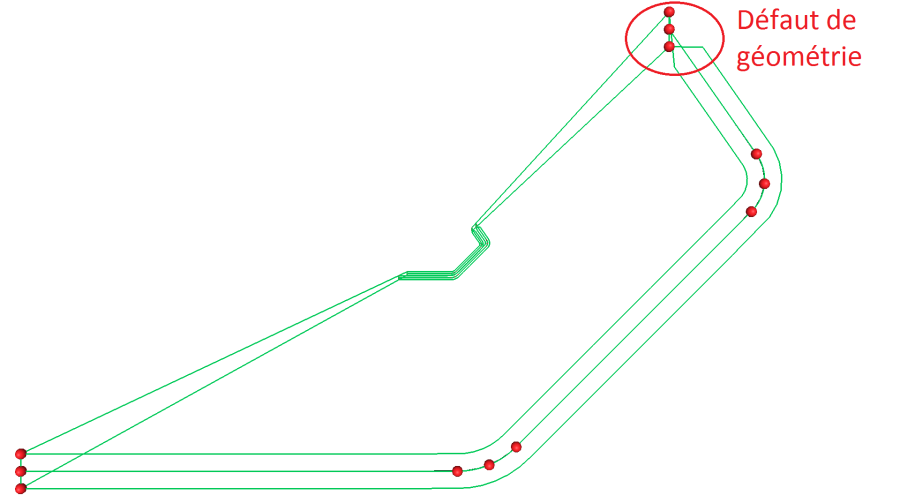 Ensuite, avec ce cas-test extrêmement réduit, j’ai pu utiliser le debugger et j’ai pu cerner le problème dans les opérateurs de projection de la polyCurve. En effet, la coordonnée réduite de chaque nœud de la polyCurve est calculée par l’opérateur de projection. Dans le cas particulier du nœud à l’extrémité de la polyCurve, la coordonnée réduite était erronée et conduisait, à son tour, à une évaluation erronée de la normale à la courbe avec pour conséquence le défaut géométrique de la rive observé. En effet, dans les routines de prise de décision de l’opérateur de projection de la polyCurve, dans le cas d’un segment frontière, j’ai observé que le type d’appartenance initialement à
Ensuite, avec ce cas-test extrêmement réduit, j’ai pu utiliser le debugger et j’ai pu cerner le problème dans les opérateurs de projection de la polyCurve. En effet, la coordonnée réduite de chaque nœud de la polyCurve est calculée par l’opérateur de projection. Dans le cas particulier du nœud à l’extrémité de la polyCurve, la coordonnée réduite était erronée et conduisait, à son tour, à une évaluation erronée de la normale à la courbe avec pour conséquence le défaut géométrique de la rive observé. En effet, dans les routines de prise de décision de l’opérateur de projection de la polyCurve, dans le cas d’un segment frontière, j’ai observé que le type d’appartenance initialement à BELONG_OUT passe à BELONG_IN, ce qui induit une remise à zéro de l’opérateur de projection et entre autres de la coordonnée réduite (1 → -1). C’est précisément l’origine des problèmes rencontrés.
En définitive, j’ai modifié la tolérance interne de l’opérateur de projection dans la classe ProfileBuilder (0.1 au lieu de 0). De cette façon, la projection devient BELONG_EDGE lors du test d’appartenance, l’opérateur de projection n’est plus mis à zéro et la coordonnée réduite recherchée est bien correcte.
Divers
- Le répertoire
oo_meta/mtParasolid/numisheet/est supprimé, après en avoir discuté avec Cristian. - Dans la méthode
extractBoundaryForce( std::vector< std::tuple<Element*,int,double> > &boundary, mtGeo::Group & group )de Interaction.cpp, j'ai fait une petite correction de manière à permettre la compilation sous VS2012. - Petit retour des warnings à la compilation.
Fichiers ajoutés/supprimés
Renamed : oo_meta/mtDrawables/SFScalars.h/.cpp -> oo_meta/mtDrawables/StickFScalars.h/.cpp Renamed : oo_meta/mtDrawables/SFVectors.h/.cpp -> oo_meta/mtDrawables/StickFVectors.h/.cpp Added : oo_nda/rollForming/copraCpeCre/Tube.orig.cpe/.cre Added : oo_nda/montataire/battery/__init__.py Added : oo_nda/rollForming/copraBattery/__init__.py Added : oo_nda/rollForming/copraBattery/large/ijss/__init__.py Added : oo_nda/rollForming/copraBattery/large/cChannel/copra/__init__.py Added : oo_nda/rollForming/copraBattery/large/comptesRendusMeca/__init__.py Added : oo_nda/rollForming/copraBattery/large/thesisRoBo/__init__.py Added : oo_nda/rollForming/copraBattery/minWorkingExample/__init__.py Added : oo_nda/rollForming/copraBattery/springback/__init__.py Added : oo_nda/rollForming/copraBattery/large/ijss/__init__.py
Tests ajoutés/supprimés
Added : oo_nda/montataire/battery/****ALEEASFr.py Added : oo_nda/montataire/battery/****ALEEASFrGod.py Added : oo_nda/montataire/battery/****ALEEASFrLess.py Added : oo_nda/montataire/battery/****ALESRIFrLess.py Added : oo_nda/montataire/battery/****LagL1840MarcMeshMarcBCs.py Added : oo_nda/montataire/battery/****LagL1840MarcMeshMarcBCsEAS.py Added : oo_nda/montataire/battery/****LagL2040NoBCsnY2.py Added : oo_nda/rollForming/copraBattery/bavoletMovTools.py Added : oo_nda/rollForming/copraBattery/U6Short.py Added : oo_nda/rollForming/copraBattery/U6SymL.py Added : oo_nda/rollForming/copraBattery/U6SymLR.py Added : oo_nda/rollForming/copraBattery/U6SymR.py Added : oo_nda/rollForming/copraBattery/U6SymRmm.py Added : oo_nda/rollForming/copraBattery/large/cChannel/copra/cChannel.py Added : oo_nda/rollForming/copraBattery/large/cChannel/copra/cChannelALE.py Added : oo_nda/rollForming/copraBattery/large/comptesRendusMeca/bavolet.py Added : oo_nda/rollForming/copraBattery/large/comptesRendusMeca/bavoletALE.py Added : oo_nda/rollForming/copraBattery/large/ijss/bavoletALE_1.py Added : oo_nda/rollForming/copraBattery/large/ijss/bavoletALE_2.py Added : oo_nda/rollForming/copraBattery/large/ijss/tubeALE_1.py Added : oo_nda/rollForming/copraBattery/large/ijss/tubeALE_2.py Added : oo_nda/rollForming/copraBattery/large/ijss/u6ALE.py Added : oo_nda/rollForming/copraBattery/large/thesisRoBo/bavoletALE.py Added : oo_nda/rollForming/copraBattery/large/thesisRoBo/channel.py Added : oo_nda/rollForming/copraBattery/large/thesisRoBo/KitrackALE.py Added : oo_nda/rollForming/copraBattery/large/thesisRoBo/traverseALE.py Added : oo_nda/rollForming/copraBattery/minWorkingExample/U6.py Added : oo_nda/rollForming/copraBattery/minWorkingExample/U6ALE.py Added : oo_nda/rollForming/copraBattery/springback/U6ALE_1.py Added : oo_nda/rollForming/copraBattery/springback/U6ALE_2.py Added : oo_nda/rollForming/copraBattery/springback/U6SymLR_1.py Added : oo_nda/rollForming/copraBattery/springback/U6SymLR_2.py Renamed : oo_nda/rollForming/copraTests/copraLarge/ -> oo_nda/rollForming/copraTests/large/ Added : oo_nda/rollForming/copraTests/large/cChannel/copra/cChannelALEPrepro.py Added : oo_nda/rollForming/copraTests/large/ijss/bavoletALE_1.py Added : oo_nda/rollForming/copraTests/large/ijss/bavoletALE_2.py Added : oo_nda/rollForming/copraTests/large/ijss/tubeALE_1.py Added : oo_nda/rollForming/copraTests/large/ijss/tubeALE_2.py Added : oo_nda/rollForming/copraTests/large/ijss/u6ALE_1.py Added : oo_nda/rollForming/copraTests/large/ijss/u6ALE_2.py Moved : oo_nda/rollForming/copraTests/copraLarge/bavoletALE.py -> rollForming/copraTests/large/thsesisRobo/bavoletALE.py Moved : oo_nda/rollForming/copraTests/copraLarge/channel.py -> rollForming/copraTests/large/thsesisRobo/channel.py Moved : oo_nda/rollForming/copraTests/Kitrack.py -> rollForming/copraTests/large/thsesisRobo/KitrackALE.py Moved : oo_nda/rollForming/copraTests/copraLarge/traverseALE.py -> rollForming/copraTests/large/thsesisRobo/traverseALE.py
— Yanick Crutzen 2017/03/13